目的
- RDKitでFingerprintを活用して分子類似性を算出する方法をまとめる。この手の手法は多く存在するが状況に応じて使い分けるべきだと思っている。
- そこで今回は各手法の結果を図示することでおおよその傾向を掴むことを目的とする
参考
以下の1の参考文献の方のコードをほぼそのまま取ってきて実行しただけに近いページです。ちゃんと知りたい方はそちらを訪問することを強くおすすめします。
全体の流れ
- 探索用の分子構造を用意する
(今回はプロトンポンプ阻害薬の構造を使う。環構造・共有結合性の硫黄原子など様々な要素を含んでおり、各種指標の違いを把握する上で良さそうだと考えたため。) - 上記分子構造を元にして化合物データベースを探索。今回はEnamine discovery diversity setを用いる。このデータベースは化合物数は少ないものの多様な原子を含んでおり, 今回のようなテストには適している。
- 各種Fingerprintにより類似度を算出。ランク付けし上位10個を抜き出し図示化
- 構造を眺めることでそれぞれの手法の特徴がわかる(ことを望む)
コードはJupyer notebookで実行しているので各コードを上からコピペして実行するだけでお試しできるはず。
今回の探索キー(プロトンポンプ阻害薬)
from rdkit import Chem, DataStructs
from rdkit.Chem import Draw
from rdkit.Chem.Draw import IPythonConsole
from rdkit.Chem import AllChemCode language: JavaScript (javascript)proton_pump_inhibitor = Chem.MolFromSmiles("CC1=CN=C(C(=C1OC)C)CS(=O)C2=NC3=C(N2)C=C(C=C3)OC")
proton_pump_inhibitorCode language: JavaScript (javascript)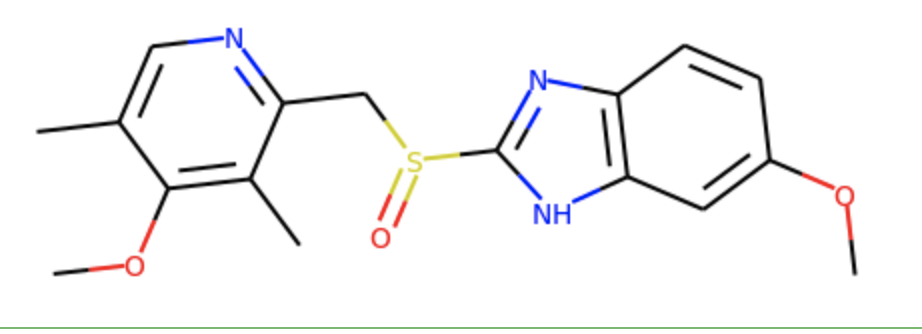
各手法の適用
まずデータベースがsdfなのでsmilesに変換した。ネット上を探せばスクリプトが落ちてると思うので変換は各自でお願いします。
変換した上でmolオブジェクトをsmilesから生成する。
# Generate a list of mol objects from the database
mols = []
with open("Enamine_Discovery_Diversity_Set_plated_10240cmds_20200806.smiles", "r") as f:
for smiles in f:
mols.append(Chem.MolFromSmiles(smiles))Code language: PHP (php)次にmolオブジェクトおよびfingerprintを入力としてfingerprintの値が上位10個の分子を表示する関数を作っておく。
def generate_figures_of_top10_molecules(set_of_mol_and_fps):
mol_and_fps_sortedby_fps = sorted([line for line in set_of_mol_and_fps], key = lambda x:-x[1])
top_ranked_mols = [ele[0] for ele in mol_and_fps_sortedby_fps][0:10]
return Draw.MolsToGridImage(top_ranked_mols, molsPerRow=5, subImgSize=(300,300), legends = ['{:.3f}'.format(ele[1]) for ele in mol_and_fps_sortedby_fps][0:10])Code language: JavaScript (javascript)MACCS
概要
- MDA社により開発
- 例えば以下のような基準で類似性を判定
- Are there fewer than 3 oxygens?
- Is there a S-S bond?
- Is there a ring of size 4?
- Is at least one F, Cl, Br, or I present?
- 本来900個ほど記述子があるらしいが一般公開されているのは166個
コード
# get MACCS for the key structure
maccs_proton_pump_inhibitor = AllChem.GetMACCSKeysFingerprint(proton_pump_inhibitor)
# get MACCS for mols in the database
maccs_fps = []
for mol in mols:
try: # to get rid of erroneous smiles
maccs_fps.append(AllChem.GetMACCSKeysFingerprint(mol))
except:
pass
# Calculate Tanimoto similaririty
maccs = DataStructs.BulkTanimotoSimilarity(maccs_proton_pump_inhibitor, maccs_fps)
mol_and_fps_maccs = set(zip(mols, maccs))
generate_figures_of_top10_molecules(mol_and_fps_maccs)Code language: PHP (php)計算結果
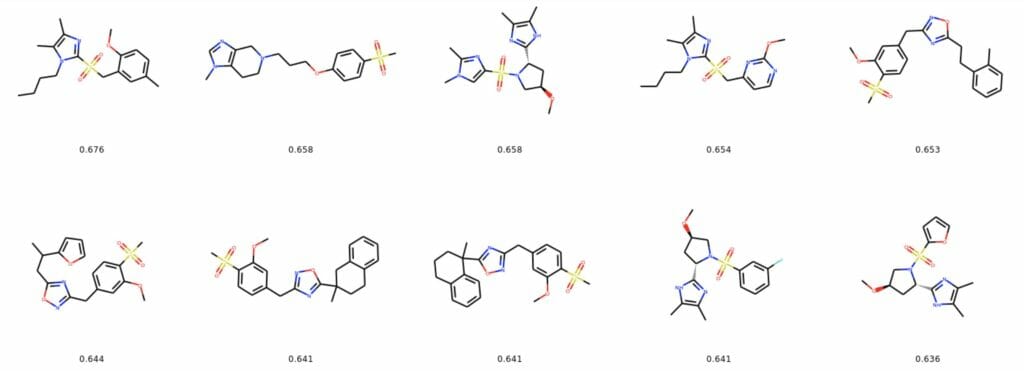
全ての分子がスルホニル基(−S(=O)2−)を含んでいる。CH3-O-のエーテルもちらほらと見られる。後者は直接探索キーに含まれており、前者は探索キーの構造に含まれるスルホキシド基(−S(=O)−)と類似性が高い。そのためこの手法は官能基の一致を高く評価していると考えられる。
Topological fingerprint
概要
- 正直よくわかっていないのだが, 原子ごとに結合次数の情報を保存している…らしい。
コード
# get rdkit fingerprint for the key structure
rdkit_proton_pump_inhibitor = Chem.Fingerprints.FingerprintMols.FingerprintMol(proton_pump_inhibitor)
# get rdkit fingerprint for mols in the database
rdkit_fps = []
for mol in mols:
try: # to get rid of erroneous smiles
rdkit_fps.append(Chem.Fingerprints.FingerprintMols.FingerprintMol(mol))
except:
pass
rdkit = DataStructs.BulkTanimotoSimilarity(rdkit_proton_pump_inhibitor, rdkit_fps)
mol_and_fps_rdkit = set(zip(mols, rdkit))
generate_figures_of_top10_molecules(mol_and_fps_rdkit)Code language: PHP (php)計算結果
先程の結果とは異なり官能基の一致度は低い。
言語化するならば「カルボニル基を含む炭素鎖により連結された一つ以上の環状構造」といったところだろうか。この結果からもtopological fingerprintが結合情報を利用するタイプのfingerprintであることが見て取れる。結合次数が重要な場合には有用なフィンガープリントだろう。
Morgan finger print
概要
- これがわかりやすい。特定の原子から特定の距離内(radius)に存在する原子を取得し, それらの特徴をidentifierで記述。そしてhash functionで特定のbit長を持つバイナリ値に押し込めるというイメージ(間違っているかもしれません)
コード
#Morgan fingerprint
morgan_proton_pump_inhibitor = AllChem.GetMorganFingerprintAsBitVect(proton_pump_inhibitor, 2, 2048)
# get rdkit fingerprint for mols in the database
morgan_fps = []
for mol in mols:
try: # to get rid of erroneous smiles
morgan_fps.append(AllChem.GetMorganFingerprintAsBitVect(mol, 2, 2048))
except:
pass
morgan = DataStructs.BulkTanimotoSimilarity(morgan_proton_pump_inhibitor, morgan_fps)
mol_and_fps_morgan = set(zip(mols, morgan))
generate_figures_of_top10_molecules(mol_and_fps_morgan)Code language: PHP (php)fingerprintに使うbit数が少なすぎると問題らしいので。今回は先駆者様を参考にbit数は2048, そしてradiusは2と4の2つを試してみる。
計算結果
まずradius = 2から。この手法はsparseなベクトルになるので, Tanimoto scoreは前述のものより低く出る。分子骨格の一致度が前述の2つの方法より高い。また窒素を含む環状化合物である点もよく再現されている。
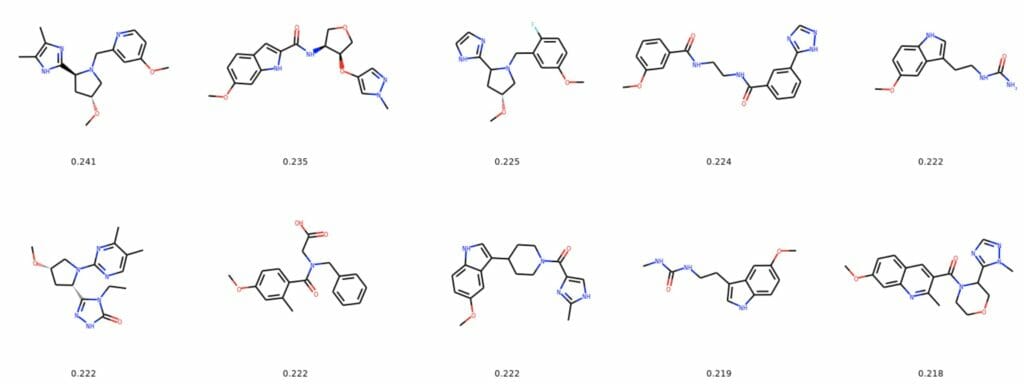
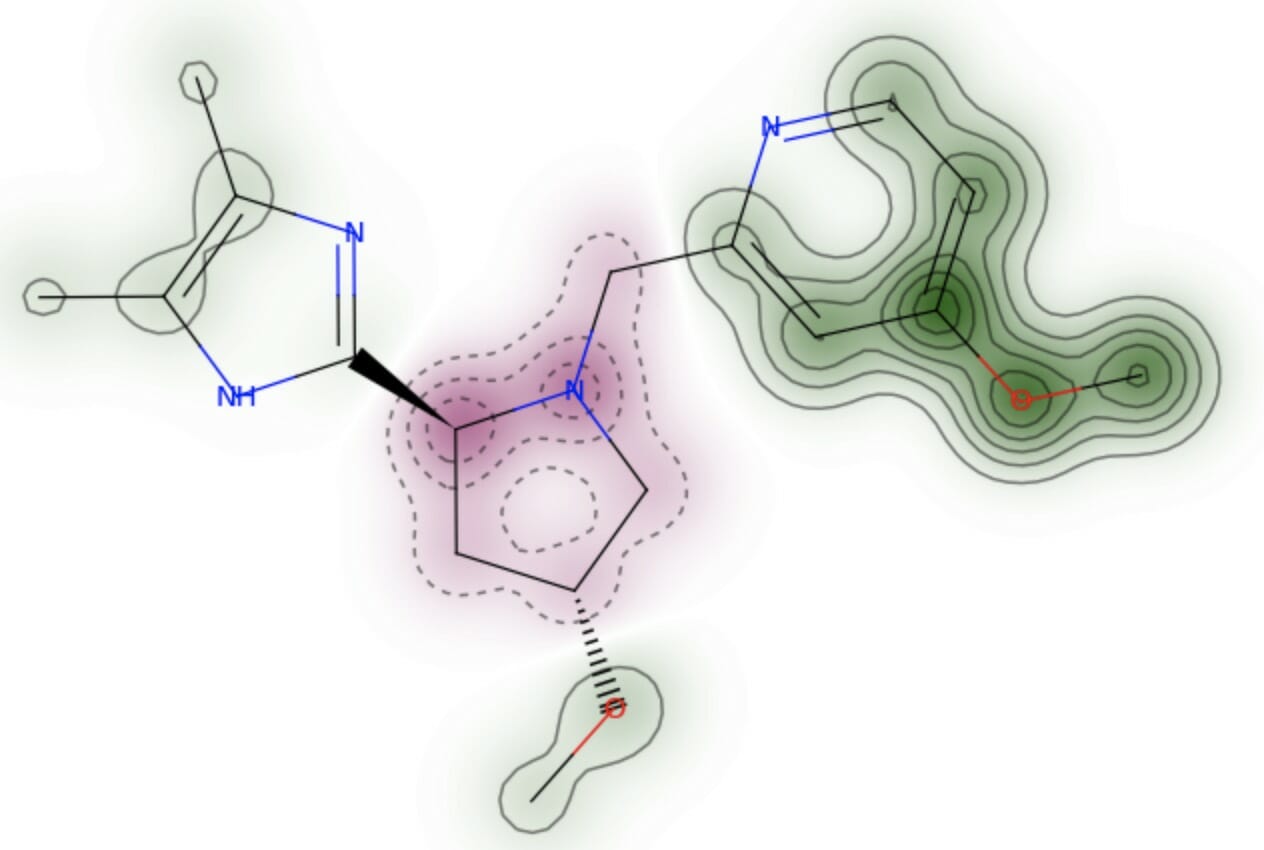
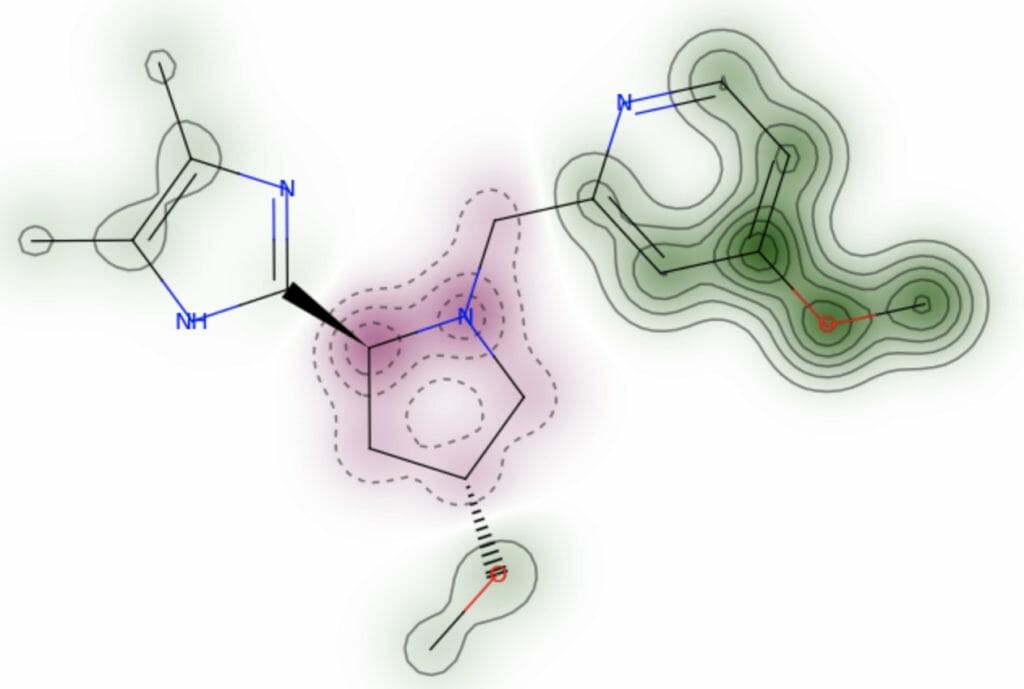
上図の中で一番tanimoto scoreが高い分子と検索キーとの類似度を以下に示す(緑色:類似度高, ピンク:類似度低)。キー構造に存在する環構造およびO-CH3部位で類似度が高く出ている。
次にradius = 4。radius = 2では環状構造を1つしか含まない構造が見られたが, radius = 4では全ての分子がキー構造と同様に複数の環構造を持つようになった。これはradius = 4ではより遠距離までの構造評価がされるようになった結果, キー構造中の「直鎖構造で隔てられた複数の環構造が存在する」という情報が取り込まれるようになったからだろう。
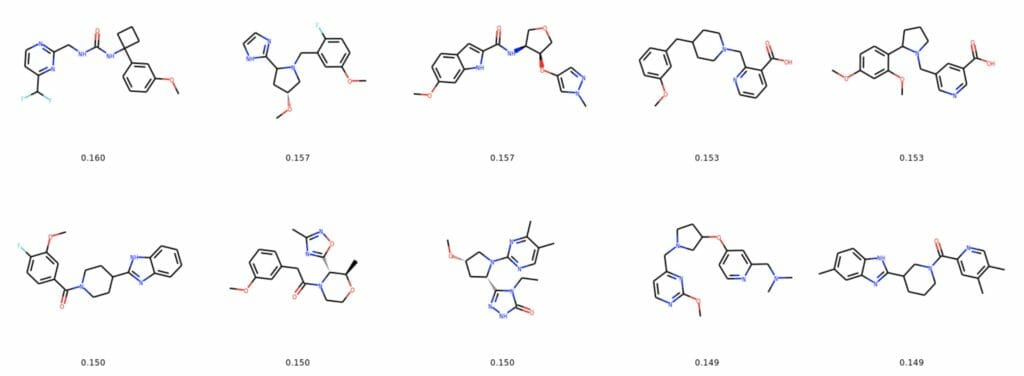
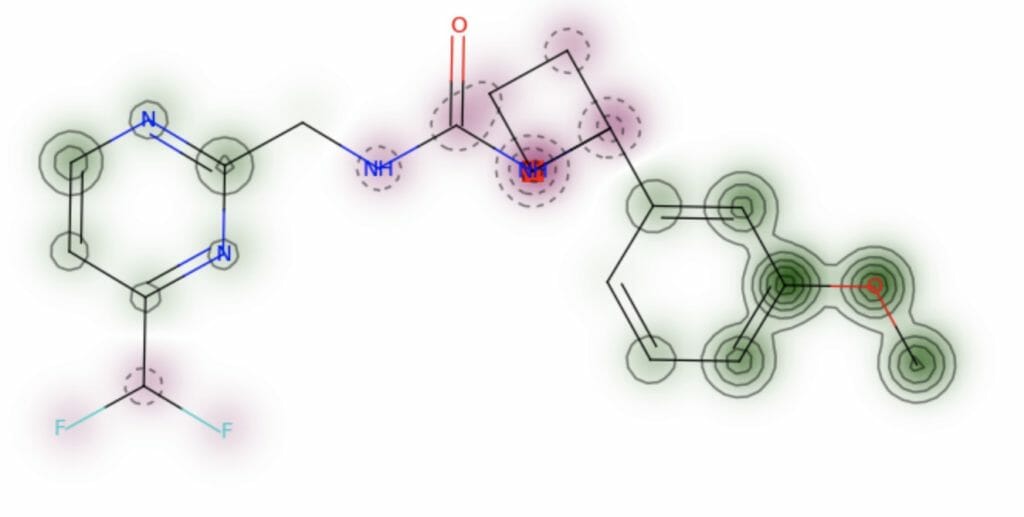
radius = 4でも同様にtop分子のどの部分がキー構造と似ていると判断されたかを以下に図示。傾向はradius = 2のときと同じで環構造およびO-CH3部位で類似度が高く出ている。
Atom pair fingerprint
概要
- 先程のmorgan fingerprintが一定の距離半径内の原子のみを考えていたのに対し, こちらは全ての原子ペアを考慮して計算するためより遠距離の関係性も取り込める。
コード
#Atoppair fingerprint
atompair_proton_pump_inhibitor = Pairs.GetAtomPairFingerprintAsBitVect(proton_pump_inhibitor)
# get rdkit fingerprint for mols in the database
atompair_fps = []
for mol in mols:
try: # to get rid of erroneous smiles
atompair_fps.append(Pairs.GetAtomPairFingerprintAsBitVect(mol))
except:
pass
atompair = DataStructs.BulkTanimotoSimilarity(atompair_proton_pump_inhibitor, atompair_fps)
mol_and_fps_atompair = set(zip(mols, atompair))
generate_figures_of_top10_molecules(mol_and_fps_atompair)Code language: PHP (php)計算結果
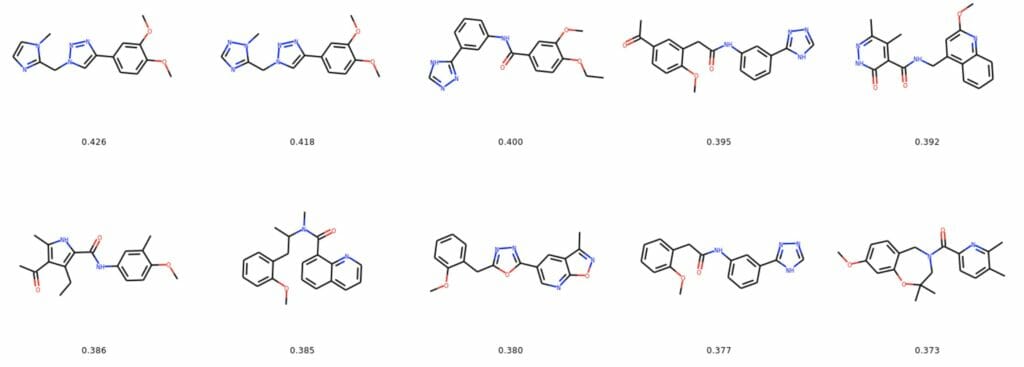
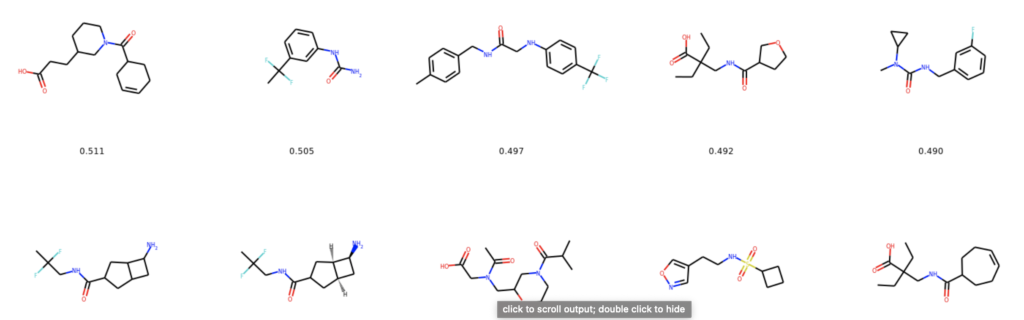
radius = 4の時のMorgan fingerprintと並んで構造は似ているように見える。構造が重要だと考えているならばMorganやAtom pairは良い手法なのかもしれない。但し共有結合性を発揮するために重要な硫黄原子はいずれの結果にも含まれていなかった。
まとめ
- いずれの方法も一長一短である。MACCSは官能基重視, その他は全体的な構造重視といった感じか。Topological fingerprintは精度が低そうなのでAtom pair fingerprintかMorgan finger printでいいんじゃないかという気がする。
- 共有結合性の阻害剤をキーとして探索開始したにも関わらず, MACCS以外では硫黄を含む化合物がほとんどヒットしていない。こうした特定の官能基が重要だと考える場合はMCSを用いた部分構造探索をしないとおそらくダメなんだろうと思う。
- というわけで次回はMCSについて書く。