目的
- AutoDockを用いたドッキング計算方法の流れを理解する
なぜAutoDock?
- 以前に「AutoDock Vinaでドッキング計算をする」という記事でAutoDock Vinaの使い方を記載した。AutoDock Vinaはempiricalおよびknowledge-basedなスコアリング関数により高速かつ精度良くな計算が可能である。
- 一方で強い電荷を持つ系では前述の経験的なスコアリング関数がうまく相互作用を記述できない可能性がある(らしい)。このような系では静電相互作用をクーロン力に基づいて計算するほうが良い結果を与える可能性がある。
- AutoDockはForce-fieldに基づくドッキングソフトウェアで, AutoDock Vinaと同様によく用いられている。
- 本記事はAutoDockでの計算方法を理解することを目的とする。
参考
- チュートリアル(公式のチュートリアルは更新日が2009年と古いのでこちらのほうが良いと思う):https://sites.ualberta.ca/~pwinter/Molecular_Docking_Tutorial.pdf
- 理論的背景:http://autodock.scripps.edu/faqs-help/tutorial/using-autodock-4-with-autodocktools/UsingAutoDock4WithADT.ppt.Handouts3pp.v3.pdf
必要なツールのセットアップ
必要なツールは本来Pymol, Autodock4, Autodock MGL toolsの3つ…なのだがAutodock MGL toolsのGUIはAutoDock Vinaの時と同様にMac OS Catalinaで動かない。
AutoDock Vinaの時と異なり, AutoDockに対応したサードパーティのソフトウェアが無さそうなのでDockerを使って無理やりAutoDock ToolsをMac上で動かすことにした。
AutoDock4
開発元からダウンロードした。ダウンロードには登録が必要。商用利用も可能なようだがライセンス関係はさっぱりわからないので各自で調べて頂きたく。
Chimera
Chimera本体をインストーラからインストールする。
Pymol
元々Macに入っていたので割愛。
AutoDock MGL tools
Linuxを使っている人だったら簡単にインストールできるのだと思う。私はMac OSX Catalinaを使っているせいでGUIが動かないのでLinux版をDocker上で動かすことにした。
計算の実施
計算の流れは大まかに以下のようになる。最初だけAutoDock Vinaと一緒だがあとは大きく異なる。AutoDock vinaと違って親切設計じゃないのだ。
- まずligandとreceptorに水素原子と電荷を付加し, pdb形式で書き出す。
- Grid parameter fileを作る(.gpfファイル)
- AutoGrid4(解凍済みのAutoDockのフォルダに入っている)を用いてマッピング情報を生成。
- Docking parameter fileを作る(.dpfファイル)
- AutoDockを実施
1. (Pymol) 計算対象のリガンドおよびタンパク質をPDB形式で書き出す
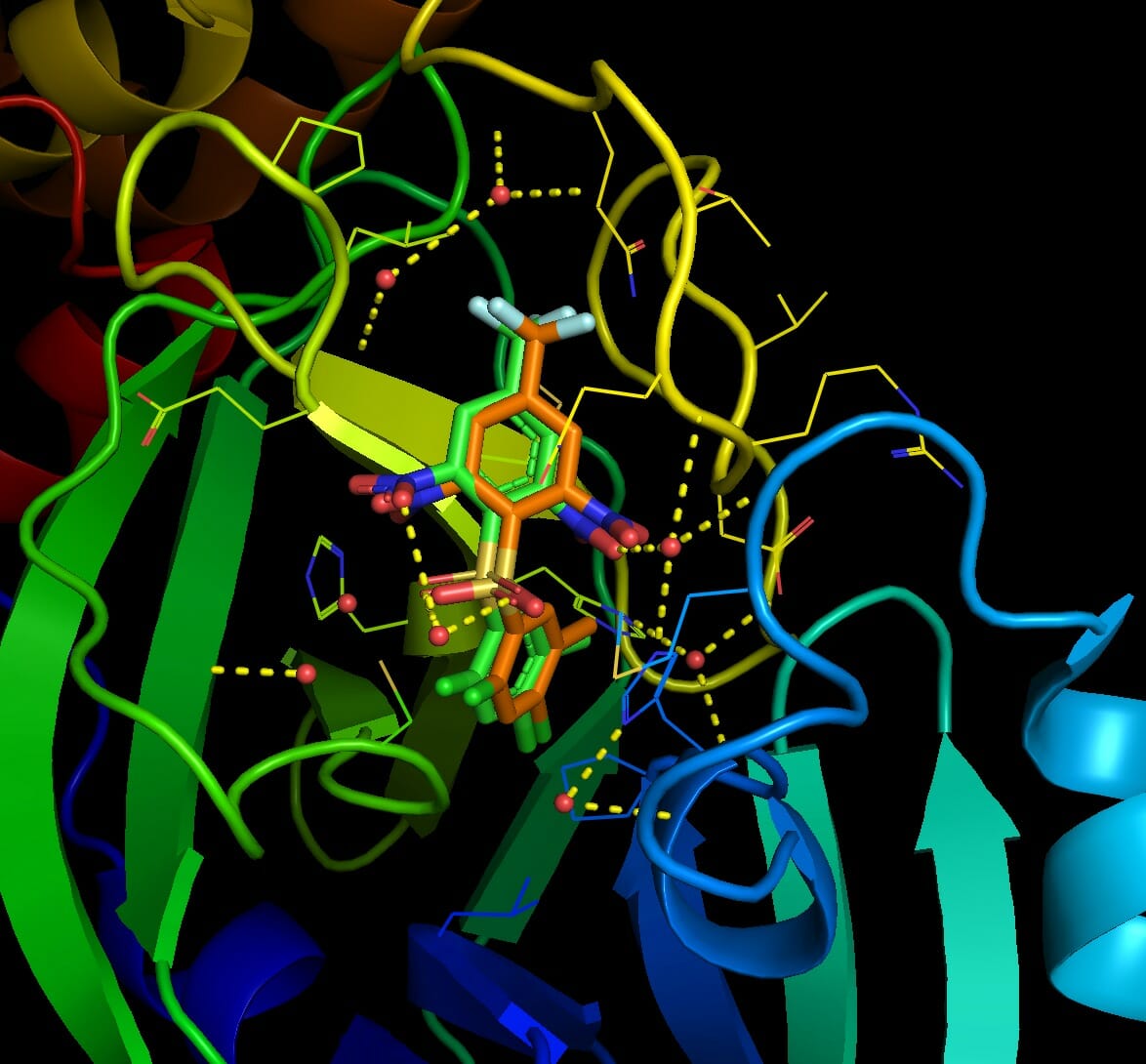
AutoDockの計算結果をAutoDock Vinaの結果と比較したいので, 前回の記事と同様にSARS-Cov-1のMain protease(PDB ID: 2GZ7)を計算対象にした。タンパク質の前処理は以前の記事と同様なので前回の記事を参照いただきたい。
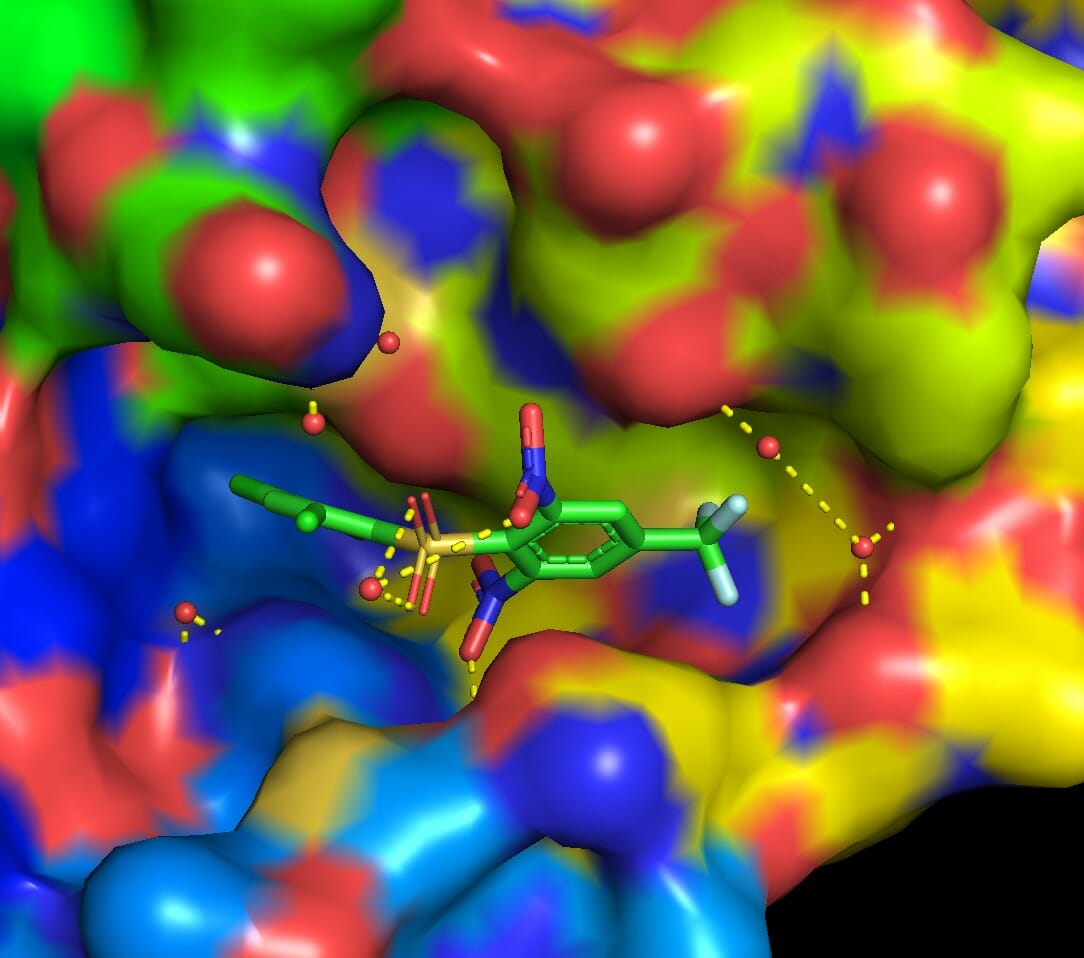
2~4. (MGL Tools) 計算対象のタンパク質とリガンドに前処理を実施
ここからが大分異なる。基本的にはチュートリアルを参照いただきたい。基本的に流れに沿っていけばシミュレーションはできるはず。以下には要約だけ記す。
- Grid parameter fileを作る(.gpfファイル)
- AutoGrid4(解凍済みのAutoDockのフォルダに入っている)を用いてマッピング情報を生成。
- Docking parameter fileを作る(.dpfファイル)
- AutoDockを実施
5. (AutoDock) ドッキングを実施
前処理が完了したら以下のコマンドでAutoDockを実行する。
/(path_to_autodock)/autodock4 –p autodock.dpf –l autodock.dlg &計算結果はdlgファイルとして得られる。pdbqtファイルに変換するには以下のコマンドを用いる(参考)。pdbqtファイルはPymolで可視化できる。
grep '^DOCKED' my_docking.dlg | cut -c9- > my_docking.pdbqtCode language: JavaScript (javascript)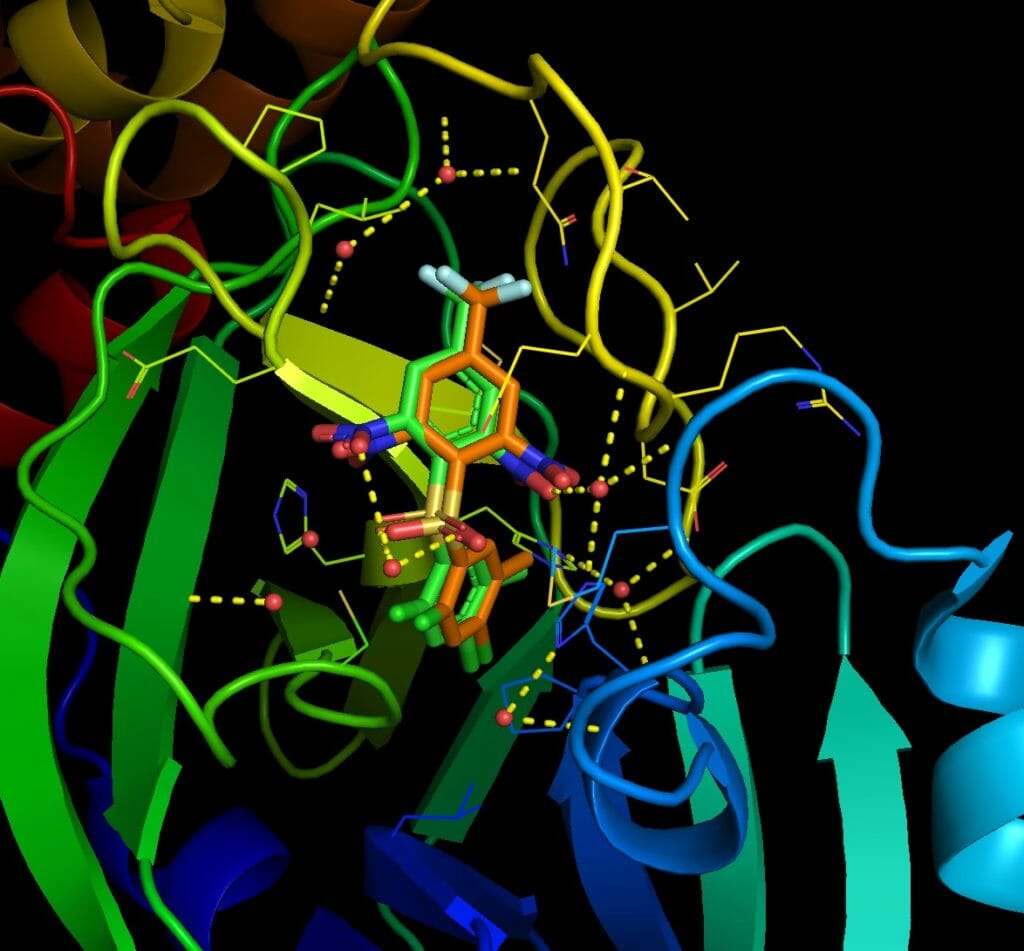
図から結晶構造を精度良く再現できていることがわかる。今回用いたリガンドは大きな電荷を持たないのでAutoDock VinaでもAutoDockでも大きな差異は生じていない。分極の大きいATPあたりを結合させると差異がもしかしたら明確になるかも知れない。
さて, ここまで見てきたようにAutoDockではグリッドを切ったり入力ファイルを作ったりといった前処理が煩雑である。pdbqtファイルを作ってボックスサイズを定義するだけでシミュレーションが可能なAutoDock vinaと比べると体感で5倍くらいはめんどくさい。
従ってAutoDock Vinaで十分精度が出る系だったら, 計算が遅くて設定がめんどくさいAutoDockをわざわざ使う意味はあまりないんじゃないかと思う。電荷の大きな系などではAutoDock VinaとAutoDockを併用してどちらの精度が上か検討するのが良いだろう。